
Sobre o artigo de Klotz
& Rintelen 2014 - Identidade do Caridina
“Crystal/Bee”
Introdução
– identificação de espécies através de testes genéticos
Ferramentas
moleculares de identificação foram desenvolvidas inicialmente para estudar
relações entre indivíduos, desde simples Testes de Paternidade até estudos de
árvores filogenéticas entre espécies. Uma aplicação direta desta tecnologia é a
identificação de espécies baseada em amostras teciduais. Tem extenso uso em
veterinária forense, identificando espécies a partir de carcaças ou pequenas
amostras de sangue e tecidos. Um exemplo interessante é a identificação de
espécies protegidas e ilegais de baleias a partir de amostras de carne
comercializada em mercados asiáticos.
As técnicas mais
tradicionais utilizavam testes imunes e eletroforese de proteínas, mas têm sido
cada vez mais substituídos por novas ferramentas usando ácidos nucleicos, com
uma série de vantagens em termos de sensibilidade, especificidade e acurácia.
Além da precisão muito superior, a análise pode ser feita a partir de amostras
pequenas, mais degradadas, ou fixadas em coleções.
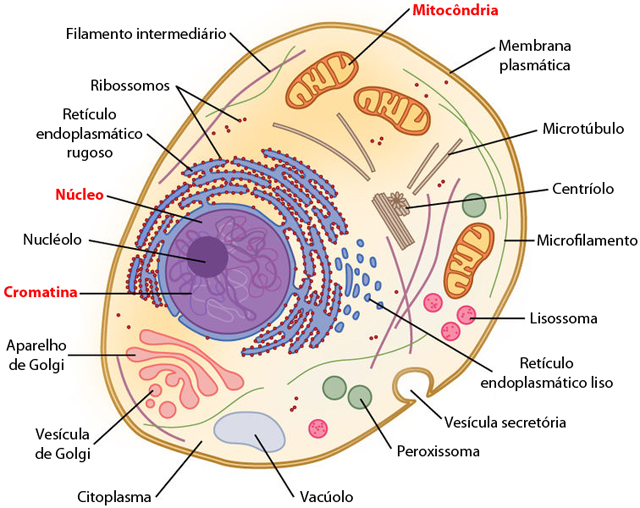
Estruturas de uma típica célula eucariótica, destaque para Mitocôndria e Núcleo celular. Ilustração de CFCF para Anatomy & Physiology, Connexions Web site. Licença Creative Commons. Arquivo original pode ser visto aqui
DNA
mitocondrial e DNA nuclear, Microssatélites
Quando se pensa em
DNA no interior da célula, imediatamente vem à mente o material genético
encontrado no núcleo celular. Na reprodução sexuada, há fusão do DNA nuclear
(nDNA) do espermatozóide e do óvulo, por exemplo em seres humanos, os 23
cromossomos do pai e os 23 da mãe se combinam, totalizando os 46 cromossomos
das nossas células. Porém, existe outro local dentro da célula onde existe DNA:
na mitocôndria. Possui um sistema próprio de replicação, transcrição e
translação.
Com poucas exceções,
as células de espécies eucarióticas possuem mitocôndrias, uma diminuta organela
envolta por uma membrana própria, que gera boa parte do ATP (fonte de energia)
da célula. É uma organela muito especial, porque é a única que possui um
material genético próprio. Acredita-se que a mitocôndria seja um vestígio de
uma bactéria endosimbionte que foi incorporada no citoplasma da célula
hospedeiro em algum momento do seu passado evolutivo.
Já vimos que há
combinação do nDNA paterno e materno quando há reprodução. Porém, na grande maioria
dos organismos com reprodução sexuada, o DNA mitocondrial (mtDNA) é herdado
somente da mãe. O mtDNA do pai é destruído ou não-incorporado no processo.
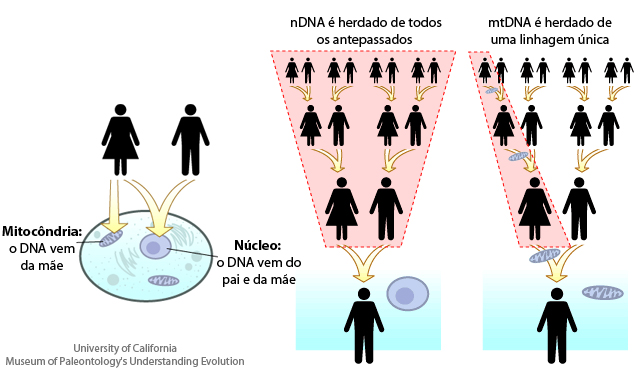
Heranças
de DNA nuclear e DNA mitocondrial. Ilustração original de University of
California, Licença Creative Commons. A página da Understanding
Evolution pode ser vista aqui
O mtDNA do filho é
idêntico ao mtDNA da mãe. Porém, ao longo de muitas e muitas gerações, o mtDNA
tem uma relativamente alta e constante taxa de mutação, com uma velocidade de
evolução acelerada. Implica em variações de sequência de graus variados que
podem ser detectados e quantificados, a fim de graduar a distância evolutiva
dentre espécies, ou mesmo indivíduos de uma mesma espécie. Estas pequenas
variações também permitem identificar locais de ramificações de ancestralidade,
e construir complexas árvores filogenéticas. Já que a taxa de mutação destes
genes é sabida, pode-se estimar também o momento quando estas bifurcações
ocorreram no passado. Por este motivo, alguns chamam o mtDNA de “relógio
molecular”.
Em 1987, o mtDNA foi
usada por cientistas da Nature para
traçar uma ancestralidade materna dos seres humanos até a África, a famosa
“Eva” mitocondrial de 200.000 anos atrás.
Em cada célula de
mamíferos, há cerca de 1700 mitocôndrias, respondendo por cerca de 1~2% do
total de DNA. Em seres humanos, há 37 genes em cada mitocôndria, e 70.000 genes
nucleares em cada núcleo celular. Embora a quantidade total de genes do nDNA
seja muito maior, e, neste sentido, a identificação de espécies seja mais
precisa usando-se esta ferramenta, a taxa de mutação de nDNA é mais baixa,
espécies diferentes mas próximas podem ter muitos loci em comum, e serem indistinguíveis.
Estas diferenças
entre nDNA e mtDNA são bastante interessantes, e trazem vantagens e
desvantagens para análises isoladas dos dois tipos de material genético.
Geralmente são usadas preferencialmente mtDNA, mais simples, facilitando
bastante a interpretação dos resultados de árvores genéticas. Em comparação ao
nDNA, o mtDNA pode ser isolado de amostras de cabelo, de amostras teciduais
degradadas e escassas, já que possui muitas cópias por célula.
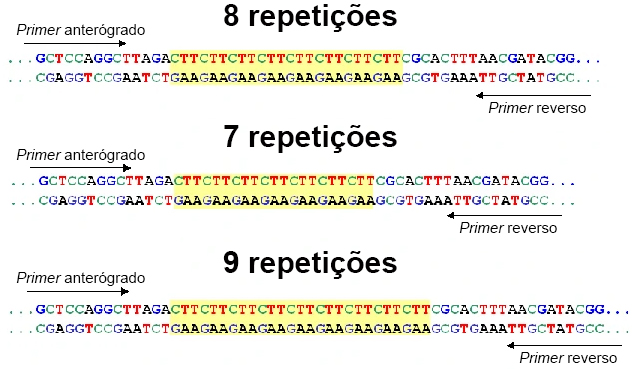
Exemplos de Microssatélites. Figura original de University of Vermont, Ecology and Management of Invasive Species, Licença Creative Commons. O arquivo original pode ser visto aqui
Trabalhos de sequenciamento de genes humanos nos anos 80 mostraram um fato curioso, a presença de múltiplas repetições de sequências curtas de pares de bases, tipicamente em agrupamentos de 5 a 50 cópias. Estas fileiras são chamadas de Microssatélites (um nome vindo do aspecto em faixas periféricas na centrifugação do genoma) ou STRs (Short Tandem Repeats). Sua função ainda é motivo de debate, são bem mais comuns em áreas não codificadoras, e estas regiões possuem uma taxa de mutação mais elevada, sendo desta forma úteis em estudos filogenéticos. Uma espécie de "impressão digital genética", possuem ainda ampla utilização em testes de paternidade, Botânica, Oncologia, Medicina Forense, como curiosidade, a identificação do criminoso nazista Josef Mengele foi baseada essencialmente nesta ferramenta.

Caridina logemanni, foto de Chris Lukhaup.
Metodologia
do trabalho dos Drs. Klotz e Rintelen
Em 2014, os Drs. Werner Klotz e Thomas von Rintelen publicaram
na revista Zootaxa o artigo
intitulado To “bee” or not to be — on some
Ornamental Shrimp from Guangdong Province, Southern China and Hong Kong SAR,
with Descriptions of Three New Species. O trabalho é muito rico, com
descrições de novas espécies, seus estágios larvares, relações filogenéticas, etc.
Além da repercussão no meio científico, o artigo foi bastante comentado em
meios aquarísticos (grupos de discussão, fóruns, sites, blogs), pela descrição de duas novas
espécies: a primeira é o Caridina mariae,
que é o “Tiger Shrimp” criado há muito tempo em aquários, mas cuja espécie não
havia sido identificada ainda. A segunda é a Caridina logemanni, que foi interpretada como sendo a espécie de
aquários “Bee Shrimp” ou “Crystal Shrimp”, porém, com algumas discordâncias nas análises genéticas, suscitando dúvidas.
De fato, os autores
fizeram uma interessante descoberta, uma nova espécie de camarão que tem o
aspecto idêntico aos “Bee” selvagens tradicionalmente criados em aquários,
batizada de Caridina logemanni. Existem diversas outras espécies com faixas
escuras e claras largas (“padrão Bee”, como o C. trifasciata, C. maculata,
C. venusta e Paracaridina zjinica, inclusive com mutações vermelhas), mas a que
mais se parece com o “Bee” selvagem é o C.
logemanni. Foi coletado somente em três pequenos córregos montanhosos na
região dos Novos Territórios, Hong Kong. Nenhuma outra espécie de camarão vivia
junto com eles, mas grandes populações de C.
cantonensis, uma espécie com ampla distribuição na região, eram encontradas
em riachos próximos.
Apesar de serem
espécies bastante próximas filogeneticamente, o aspecto das duas difere
bastante: O C. logemanni tem o típico
aspecto de “Bee”, com várias faixas largas de cor marrom enegrecida na carapaça
e abdômen, enquanto o C. cantonensis
não tem faixas, somente um corpo homogêneo translúcido com pequenas manchas
escuras (veja fotos). E certamente trata-se de espécies distintas, havia distância genética
suficiente para distingui-la da espécie-irmã Caridina cantonensis.
A distância-p entre as duas espécies foi de 2,0-3,9% (16S) e 4,5-4,9%
(COI), não muito alta, mas suficiente para caracterizá-la como outra
espécie.
Foram realizadas
também análises genéticas para a identificação das diversas espécies. Excetuando-se
as Paracaridinas, Tigers e grupos de
controle, foi realizada a análise molecular de 53 espécimes de Caridina, na sua maioria de exemplares
selvagens coletados, mas que incluíam também 9 camarões ornamentais (Dr. Klotz, com. pess. 21/10/2019):
- Dois camarões comprados em lojas chinesas de aquarismo.
- Dois camarões de uma importadora alemã (selvagens "Diamond Bee" coletados em Hong Kong).
- Três camarões de aquaristas alemães ("Red Wine Taiwan Bee", "Snow White Ueno Line" e "Crystal Black").
- Dois camarões "New Bee" de um aquarista alemão.
Foi realizada a
análise de dois genes do mtDNA, o “16S” (que codifica o 16S rRNA, envolvido na
síntese proteica mitocondrial) e “COI” (que codifica a subunidade I da enzima
Citocromo Oxidase, envolvida na fosforilação oxidativa).
Dos 9 exemplares ornamentais
de aquário (todos com faixas, “padrão Bee”), os resultados foram (para 16S):
- 2 camarões comprados
em lojas da China: formam um clado irmão aos identificados como C. trifasciata, mas com uma distância
grande o suficiente para sugerir se tratar de uma espécie distinta.
- 2 camarões "New Bee" de um aquarista alemão: foram identificados morfologicamente e geneticamente como C. venusta.
- Um dos camarões "Diamond Bee" coletados em Hong Kong: foi identificado morfologicamente como C. logemanni, mas geneticamente se agrupa com os quatro abaixo, dentro do clado C. cantonensis.
- O outro "Diamond Bee" coletados em Hong Kong, e os três camarões ornamentais de aquaristas alemães: inseridos geneticamente no clado do C. cantonensis.
A COI não pôde ser
amplificada em vários espécimes, resultando em um conjunto de dados menor.
Somente foi possível analisar dois exemplares de aquário, um dos que formam um
clado irmão ao C. trifasciata, e um
dos inseridos no clado do C. cantonensis.
Ou seja, confirmam e reforçam os achados do 16S.

Caridina cantonensis, foto de Paul Ng Y.C.
Conclusões
do trabalho - Identidade do Caridina
“Crystal/Bee”?
Parece claro que
alguns camarões ornamentais de aquário pertencem às espécies C. venusta e C. trifasciata (ou uma espécie muito próxima a esta última). Estas
duas espécies mostram um fenótipo do tipo “Bee”, mesmo em animais coletados na
natureza. Deve-se concluir que uma parcela dos camarões “Bee” criada em
aquários no mundo devem ser destas duas espécies, especialmente os selvagens.
A grande dúvida é em
relação aos demais cinco camarões,
todos com um fenótipo “Bee”. Somente um selvagem foi identificado morfologicamente como C. logemanni, os demais têm morfologia
indeterminada, mas com um padrão “Bee”. Curiosamente, todos os seis (inclusive
aquele com morfologia de C. logemanni)
foram identificados geneticamente como C.
cantonenis, uma espécie sem faixas. Como isto é possível?
1. O
problema dos híbridos
Para o mtDNA, híbridos
possuem somente a assinatura genética da sua herança materna. Desta forma, uma
análise isolada do mtDNA não é adequada para a identificação de espécies, nos
casos onde existe a possibilidade de hibridização, como populações em zonas de
fronteira entre distribuição de duas espécies próximas, ou criação conjunta em
cativeiro de duas espécies próximas. Nestes casos, dados mitocondriais devem
ser suplementados com ferramentas moleculares baseadas em genes nucleares, que
têm herança biparental.
Na realidade, a simples
discordância entre a análise do mtDNA e nDNA (neste último, seja por análises
cromossômicas ou fenotípicas) é uma boa dica de que pode ter havido
hibridização. A famosa teoria da nossa hibridização com Neandertais é baseada
nisto. Um trabalho recente (2008) brasileiro estudou híbridos de
Tartaruga-de-pente (Eretmochelys
imbricata) e Tartaruga-cabeçuda (Caretta
caretta) usando esta metodologia. Já foi usado em grandes gatos, para
identificar híbridos (entre sub-espécies) de Leões Africanos (Panthera leo leo) e Asiáticos (Panthera leo persica), assim como
híbridos entre Tigres de Bengala (Panthera
tigris tigris) e Siberianos (Panthera
tigris altaica).
É um fenômeno chamado
de Introgressão Mitocondrial (Evolução Reticulada), ou seja, a inserção de
mtDNA de uma espécie em indivíduos de outra espécie, através de hibridização pregressa,
e o cruzamento deste híbrido com a população da espécie original. É um fato bastante
raro em crustáceos decápodes, porque nestes, o sexo feminino é o heterogamético
(WZ), e, segundo a Lei de Haldane, híbridos heterogaméticos são mais raros,
frágeis e estéreis, limitando a propagação do mtDNA de linhagem materna.
Mas é uma hipótese para se explicar o
que ocorreu no trabalho dos Drs. Klotz e Rintelen, sugerido pelos próprios autores: camarões com mtDNA de C. cantonensis e nDNA (fenótipo) “Bee”
(ou seja, não-C. cantonensis, por
exemplo de C. logemanni). São
prováveis híbridos de C. cantonensis
com outra(s) espécie(s) com faixas, como o C.
logemanni (veja
ilustração abaixo, híbrido da última linha, F4 da direita).
Dois questionamentos
interessantes, o primeiro é o seguinte: se as duas espécies eram criadas juntas
e se hibridizaram, porque não encontramos também o inverso? Fenótipo (nDNA) de C. cantonensis e mtDNA de, por exemplo, C. logemanni? A resposta é muito
simples: um fenótipo de C. cantonensis
não é um padrão “Bee”, não é atrativo do ponto de vista ornamental. Ou seja, se
estes híbridos existiram, provavelmente foram eliminadas pela simples seleção
do aquarista. Existem outras explicações possíveis, nem sempre os híbridos têm
sobrevida e fecundidade iguais, a depender de que espécie foi o pai e qual foi
a mãe. Mas a explicação mais provável é esta.

Caridina venusta, foto de Werner Klotz.
Um último aspecto importantíssimo, se houve hibridização, o esperado seria que uma parte dos camarões de aquário se agrupassem no clado C. cantonensis, e uma parte juntamente com os C. logemanni. Porém, o que se observou foi que todos os camarões ornamentais de aquário, assim como os dois Diamond Bee selvagens coletados em Hong Kong (inclusive um deles com morfologia de C. logemanni) se agruparam no clado do C. cantonensis. Nenhum se agrupou dentro do clado logemanni, um fato estranho, mas que foi elucidado em outros trabalhos discutidos adiante.

Ilustração mostrando hibridização e introgressão mitocondrial. No exemplo, o casal original (F0) foi de Caridina cantonensis, uma espécie sem faixas, e Caridina logemanni, uma típica espécie com faixas. Os híbridos do sexo feminino transmitem adiante o mtDNA da ancestral feminina original (C. cantonensis). Note que, após muitas gerações, como os dois híbridos da última linha (F4), o fenótipo da espécie inserida no cruzamento (C. cantonensis) se dilui, e é difícil de ser detectada. O F4 da direita é o exemplo no texto de fenótipo de C. logemanni e mtDNA de C. cantonensis. Ilustração de Walther Ishikawa.
2. Sim, híbridos em aquários... mas híbridos selvagens?
Em 2018 foi publicado um interessante artigo, do Dr. Lai Him Chow e colegas da Universidade Chinesa de Hong Kong, intitulado Isolation and Characterization of Polymorphic Microsatellite Loci for Caridina cantonensis and Transferability Across Eight Confamilial Species (Atyidae, Decapoda), na revista Zoological Studies. Seu objetivo era, dentre outros, avaliar a diversidade genética do C. cantonensis, assim como de espécie próximas de Atyídeos, inclusive o C. logemanni.
Embora não seja destacado no texto do artigo, o trabalho traz uma informação bastante interessante para a nossa discussão. Foram analisados o mtDNA (COI) de 9 exemplares de C. cantonensis e 29 de C. logemanni (de dois dos três riachos descritos no trabalho original de Klotz e Rintelen, 18 e 11 indivíduos de cada riacho). Adiante, uma reprodução de parte da árvore filogenética de máxima verossimilhança gerada a partir destes dados. Mais da metade (17) dos camarões selvagens C. logemanni se agruparam geneticamente no clado C. cantonensis. Houve um claro predomínio de camarões provenientes de um dos riachos, mas alguns (2 e 3) se agruparam no outro clado. Ou seja, agora, com uma análise genética de um número maior de C. logemanni (no artigo de Klotz e Rintelen foram 2 camarões, e agora 29), mais da metade deles foram identificados geneticamente como C. cantonensis!
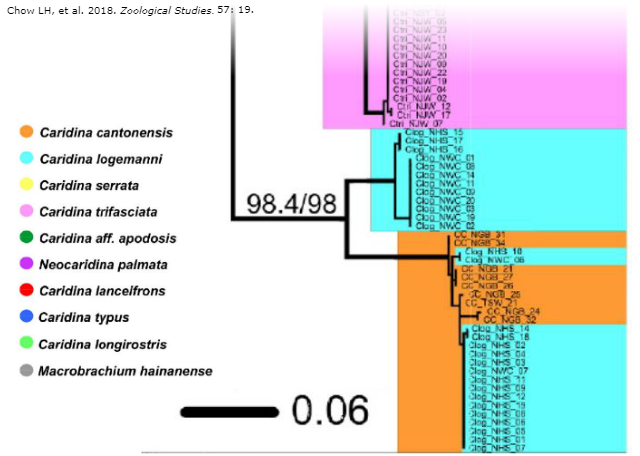
Uma parte da árvore filogenética da máxima verossimilhança gerada a partir de COI, ilustração de Chow LH, et al. Zoological Studies, 2018. 57: 19. Licença Creative Commons. Para o artigo original, clique aqui.
Este trabalho também mostrou uma grande diversidade genética do C. cantonensis, fato este já demonstrado em dois outros trabalhos (Tsang 2017 e Yam 2005). Os trabalhos mostram uma grande diferença genética entre populações de diferentes riachos, e pequena diversidade genética dentro de cada população, mesmo entre riachos separados por somente alguns quilômetros, ou tributárias de uma mesma bacia. Desta forma, uma possibilidade aventada na ocsião seria a de que o C. logemanni (e os Bees identificados como C. cantonensis) não passem de uma variante local do C. cantonensis, ou uma sub-espécie.
Este fato é lembrado pelo Dr. Klotz em um e-mail respondendo estes questionamentos, comentando também sobre outros detalhes que mostram que a conclusão não é assim tão simples (Dr. Klotz, com. pess. 23/10/2019):
"Parece que o Bee Shrimp é uma 'variante local' do C. cantonensis. Mas todos os camarões 'selvagens' dentro do agrupamento do Bee Shrimp mostram uma coloração similar que é claramente diferente do C. cantonensis da China, Hong Kong, e da porção norte do Vietnã. E a morfologia de todos os Bee Shrimp, independentemente destes serem selvagens, Crystal Red clássicos, Bee Shrimp modernos ou linhagens Taiwan foram constantemente diferentes do C. cantonensis."
"Mais tarde, eu pude coletar Bee Shrimp selvagens de dois outros córregos. Estes camarões eram semelhantes ou ao Diamond Bee Shrimp clássico (córrego 1), ou ao velho 'Color Bee' Shrimp que era vendido aqui na Alemanha nos anos 90. Estes espécimes também eram idênticos em morfologia aos Bee Shrimp do comércio de aquários, mas surpreendemente diferentes na análise 16S. Thomas explicou este fato através de introgressão causada por hibridização nos camarões do comércio, mas eu pessoalmente não acredito nisso. Por outro lado eu não tenho uma explicação clara para a topologia na árvore. Mas sabemos de situações semelhantes no gênero Neocaridina e nas espécies dos lagos centrais, onde muitas espécies são situadas erroneamente em árvores filogenéticas."
Estas dúvidas e discordâncias foram elucidadas em 2022, quando foi publicado um novo artigo também do Dr. Lai Him Chow
e colaboradores, intitulado Genetic Assessment of the Rare Freshwater Shrimp Caridina logemanni Endemic to Hong Kong and its Hybridization with a Widespread Congener, na revista Marine and Freshwater Research. Dando continuidade ao artigo anterior, seu objetivo era avaliar a diversidade genética e estrutura populacional do C. logemanni, e a extensão de hibridização com o C. cantonensis. Bastante completo, desta vez, foram utilizados um gene mitocondrial (COI), um nuclear (intron da subunidade α da enzima ATPase NaK) e seis marcadores de microssatélites. Foram analisados:
- 39 camarões C. logemanni coletados em dois riachos (duas das três populações conhecidas da espécie)
- 181 camarões C. cantonensis coletados em seis riachos próximos a estes locais
- 10 camarões com morfologia de C. logemanni comprados em lojas de aquário em Hong Kong
Um primeiro destaque é que este trabalho demonstrou de forma mais contundente que há provavelmente extensa hibridização introgressiva nas duas populações analisadas de C. logemanni, com compartilhamento de haplótipos de COI entre as duas espécies, e mistura maior de microssatélites entre-espécies do que entre-populações do C. cantonensis. E não foi encontrada uma distribuição aleatória de polimorfismo ancestral de NaK entre populações descendentes.
Desta vez, a análise de nDNA foi bastante complementar e confirmatória. Pelo COI (mtDNA) houve produção de 7 e 8 haplótipos para C. logemanni e C. cantonensis, respectivamente, e pelo NaK (nDNA) 3 e 13 haplótipos. Dois deles eram compartilhados entre as duas espécies, tanto para COI quanto NaK. Os indivíduos coletados no riacho1 pertenciam quase todos aos haplótipos de C. logemanni, tanto pelo COI quanto NaK. Boa parte dos indivíduos do riacho2 pertenciam aos haplótipos compartilhados, tanto pelo COI quanto NaK. Alguns destes haplótipos compartilhados eram dominados por indivíduos C. cantonensis. Praticamente todos os exemplares de lojas de aquarismo pertenciam a um dos haplótipos compartilhados pelas duas espécies.
Foi estimada a proporção de indivíduos híbridos nestas populações analisadas, estimando-se um número surpreendente de 66,7% para C. logemanni (84,2% em um dos dois riachos, 50% no outro), e 9,4% para C. cantonensis (até 32,5% em algumas populações). Os autores até mencionam a preocupação de inundação e erosão genética, com o desaparecimento do genoma exclusivo do C. logemanii (chamado por alguns de "extinção genética"), embora no momento ainda haja alta diversidade genética em uma das populações estudadas.
Conclusões
(agora sim!) - Identidade do Caridina
“Crystal/Bee”
Muitas espécies
selvagens têm um fenótipo “Crystal/Bee”. A identidade de exemplares selvagens
pode ser diversas espécies. Destas, a que mais lembra o padrão mais habitual
dos “Bee” selvagens é o recém descrito Caridina
logemanni. Outra possibilidade para os camarões selvagens comprados em lojas é o Caridina venusta, um fato já sugerido por alguns autores Chineses, como Wang, Liang e Li em 2008.
Camarões ornamentais "Bee" em aquariofilia também devem ser considerados como sendo Caridina logemanni. Na primeira versão deste artigo, nós alertávamos para um erro nesta identificação, devido à discordância genética, mas agora se sabe que o Caridina logemanni é uma espécie com alta porcentagem de híbridos mesmo em exemplares selvagens.
Este trabalho nos
alerta também sobre a dificuldade que pode haver na identificação de espécies
baseada somente em marcadores genéticos. A Taxonomia Genética é vista por
muitos (leigos, e até alguns biólogos) como uma ferramenta definitiva, a “palavra
final” na identificação de espécies. Vimos que não é bem assim.
Nos dias atuais,
muito se fala de uma “Taxonomia Integrada” combinando informações de múltiplas
fontes, incluindo comportamento, ecologia, geografia, morfologia e dados
moleculares. Mesmo a análise genética deve ser de multi-marcadores, sempre que
possível, como feito de forma brilhante por Lai e colaboradores em 2022.
Para bibliografia, créditos fotográficos e agradecimentos, clique aqui
Artigo publicado em 31/01/2015, última atualização em 23/12/2022